7月1日,生物学权威期刊《BMC Biology》在线发表了四川农业大学动物医学院赵兴洪/万红平团队的最新研究论文,题为“AmpHGT: expanding prediction of antimicrobial activity in peptides containing non-canonical amino acids using multi-view constrained heterogeneous graph transformer”。该研究成功构建了一种多视角约束异质图Transformer模型(AmpHGT),为高效发现含非经典氨基酸的肽类抗生素提供了新方法。

论文在线发表情况
该论文第一作者为我校动物医学院2021级本科生何永成,赵兴洪副教授为最后一位通讯作者,万红平副教授为共同通讯作者,宋旭副教授参与了该项研究。这是该团队继去年在《Nature Communications》、《Advanced Science》和《Advanced Materials》等国际顶级期刊发表新型抗菌策略相关研究成果后取得的又一重要进展。
近年来,借助机器学习与深度学习进行肽类分子设计与优化成为生物医药领域的研究热点。相比传统的蛋白质肽链,由于非核糖体抗菌肽(如多粘菌素、达托霉素和万古霉素)在结构中引入了大量非经典氨基酸,其复杂的化学组成对现有模型构成挑战。传统方法常将这类氨基酸视为“未知”(通常以“X”标记),甚至直接剔除含非经典氨基酸的序列,导致抗菌肽的活性预测准确性受限。
本研究创新性地将非经典氨基酸的化学结构信息与预训练语言模型提取的蛋白质序列特征相结合,构建多层次异质图,通过AmpHGT模型更准确地捕捉这些氨基酸的化学特性,实现对抗菌活性的有效预测。
在模型评估方面,研究首先基于经典氨基酸构建的数据集,与当前主流抗菌肽预测模型进行横向比较,验证了AmpHGT模型的性能优势。随后,研究团队从头构建了包含非经典氨基酸的训练数据集,并将AmpHGT与代表性预训练模型ESM2在测试集上进行了性能对比。结果显示,AmpHGT在非经典氨基酸数据上的预测效果优于ESM2,充分展现了图神经网络在处理复杂分子结构信息方面的潜力。
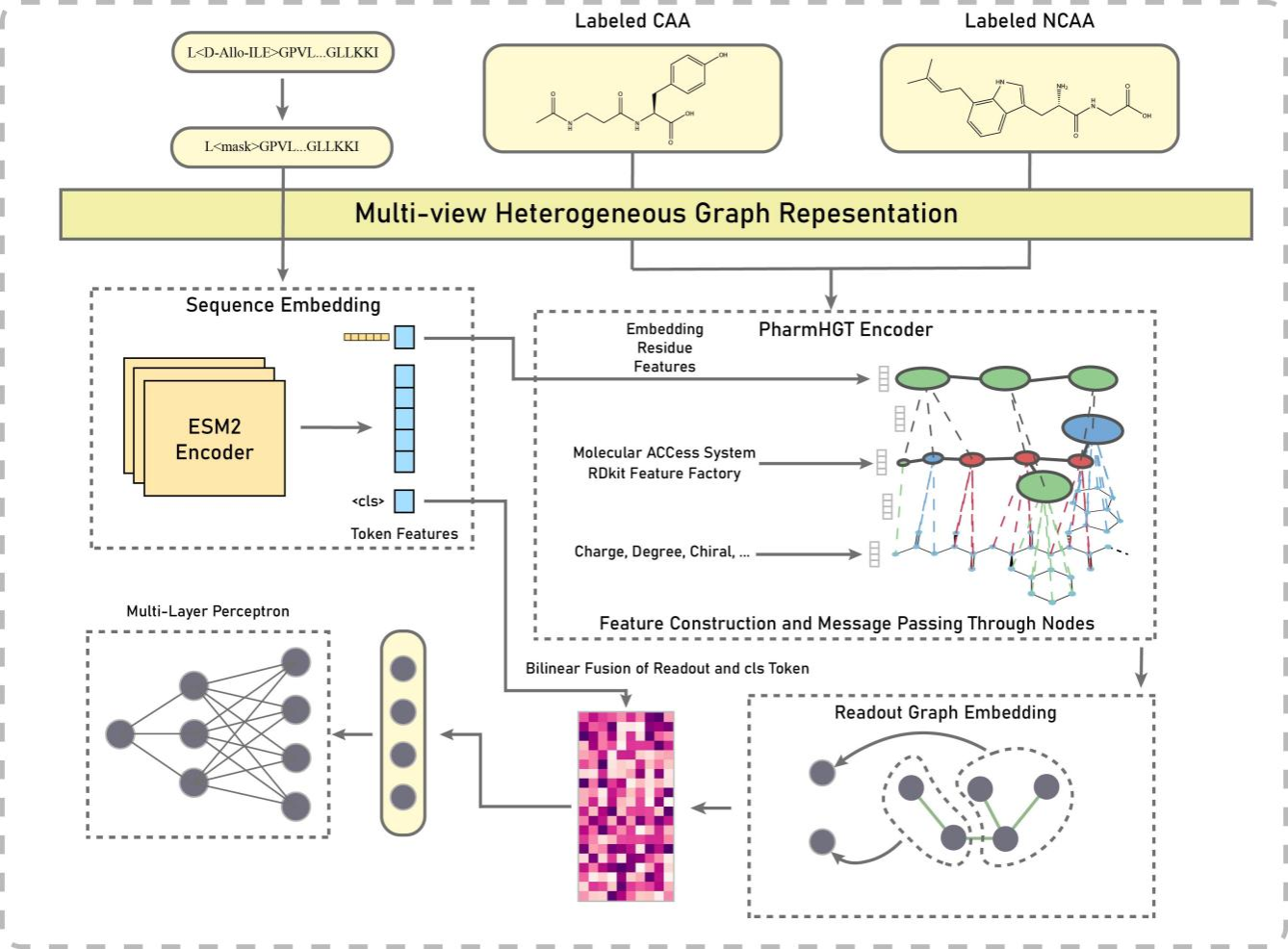
AmpHGT模型架构图
该成果为未来开发更智能、高效的抗菌肽预测及生成模型提供了坚实基础。研究工作得到了国家自然科学基金、四川省国际科技创新合作项目、四川省自然科学基金以及四川省“天府峨眉计划”等项目的资助。
原文在线地址:https://bmcbiol.biomedcentral.com/articles/10.1186/s12915-025-02253-4

